Aprenda a usar las herramientas OCR, Apache Spark y otros componentes de Apache Hadoop para procesar imágenes PDF a escala.
Las tecnologías de reconocimiento óptico de caracteres (OCR) han avanzado significativamente en los últimos 20 años. Sin embargo, durante ese tiempo, se ha hecho poco o ningún esfuerzo por combinar OCR con arquitecturas distribuidas como Apache Hadoop para procesar grandes cantidades de imágenes casi en tiempo real.
En esta publicación, aprenderá a usar herramientas estándar de código abierto junto con componentes de Hadoop como Apache Spark, Apache Solr y Apache HBase para hacer exactamente eso en un caso de uso de información de dispositivos médicos. Específicamente, utilizará un conjunto de datos públicos para convertir texto narrativo en campos de búsqueda.
Aunque este ejemplo se concentra en la información de dispositivos médicos, se puede aplicar en muchos otros escenarios donde se requiere procesamiento y persistencia de imágenes. Las compañías de seguros, por ejemplo, pueden hacer que todos sus documentos escaneados en archivos de reclamos se puedan buscar para una mejor resolución de reclamos. De manera similar, el departamento de la cadena de suministro en una planta de fabricación podría escanear todas las hojas de datos técnicos de los proveedores de piezas y hacer que los analistas puedan buscarlas.
Caso de uso:registro de dispositivos médicos
Los últimos años han sido testigos de una oleada de cambios en el campo del registro electrónico de productos farmacéuticos. El estándar ISO IDMP (Identificación de productos médicos) es uno de esos formatos de mensajes para registrar productos y las sustancias contenidas en ellos, con la identificación del producto medicinal, la identificación del empaque y la identificación del lote que se utilizan para rastrear los productos en los casos de experiencias adversas, ilegales. importación, falsificación y otros temas de farmacovigilancia. La norma exige que no solo se deban registrar los productos nuevos, sino que también se proporcione en forma electrónica la presentación más antigua/archivada de cada producto al que el público podría estar expuesto.
Para cumplir con los estándares IDMP en diferentes empresas, las empresas deben poder extraer y procesar datos de múltiples fuentes de datos, como RDBMS y, en algunos casos, hojas de datos de productos heredados. Si bien es bien conocido cómo ingerir datos de RDBMS a través de tecnologías como Apache Sqoop, el procesamiento de documentos heredados requiere un poco más de trabajo. En su mayor parte, los documentos deben ingerirse y el texto relevante debe extraerse programáticamente a escala utilizando las tecnologías de OCR existentes.
Conjunto de datos
Usaremos un conjunto de datos de la FDA que contiene todas las presentaciones 510(k) que hayan presentado los fabricantes de dispositivos médicos desde 1976. La sección 510(k) de la Ley de Alimentos, Medicamentos y Cosméticos requiere que los fabricantes de dispositivos que deben registrarse notifiquen FDA de su intención de comercializar un dispositivo médico con al menos 90 días de anticipación.
Este conjunto de datos es útil por varias razones en este caso:
- Los datos son gratuitos y de dominio público.
- Los datos encajan perfectamente con la regulación europea, que se activa en julio de 2016 (donde los fabricantes deben cumplir con los nuevos estándares de datos). Los empastes de la FDA tienen información importante relevante para obtener una vista completa de IDMP.
- El formato de los documentos (PDF) nos permite demostrar técnicas de OCR simples pero efectivas cuando se trata de documentos de múltiples formatos.
Para indexar estos datos de manera efectiva, necesitaremos extraer algunos campos de las imágenes. A continuación se muestra un documento de muestra, con los posibles campos que se pueden extraer.
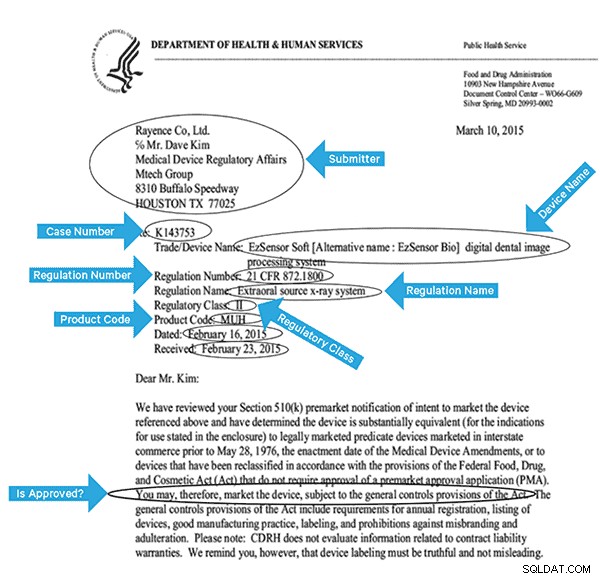
Arquitectura de alto nivel
Para este caso de uso, los archivos PDF se almacenan en HDFS y se procesan con las bibliotecas Spark y OCR. (El paso de ingestión está fuera del alcance de esta publicación, pero podría ser tan simple como ejecutar hdfs -dfs -put o usando una interfaz webhdfs). Spark permite el uso de código casi idéntico en una aplicación Spark Streaming para transmisión casi en tiempo real, y HBase es un medio de almacenamiento perfecto para acceso aleatorio de baja latencia, y es muy adecuado para almacenar imágenes, con la nueva funcionalidad MOB, para arrancar. Cloudera Search (que se basa en Apache Solr) es la única solución de búsqueda que se integra de forma nativa con HBase, lo que le permite crear índices secundarios.
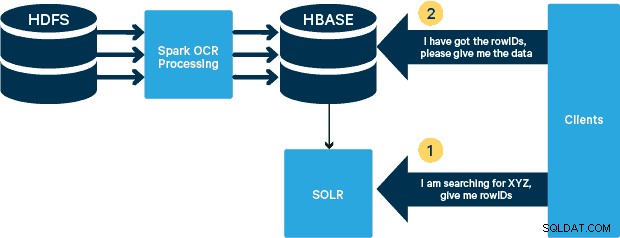
Configuración de la tabla de dispositivos médicos en HBase
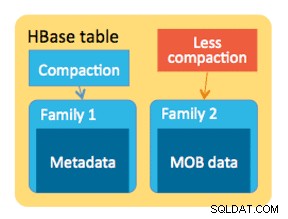
Mantendremos el esquema para nuestro caso de uso sencillo. El ID de fila será el nombre del archivo y habrá dos familias de columnas:"info" y "obj". La familia de columnas "info" contendrá todos los campos que extrajimos de las imágenes. La familia de columnas "obj" contendrá los bytes del objeto binario real, en este caso PDF. El nombre de la tabla en nuestro caso será "mdds".
Aprovecharemos la funcionalidad HBase MOB (objeto medio) introducida en HBASE-11339. Para configurar HBase para manejar MOB, se requieren algunos pasos adicionales, pero, convenientemente, las instrucciones se pueden encontrar en este enlace.
Hay muchas formas de crear la tabla en HBase mediante programación (API de Java, API REST o un método similar). Aquí usaremos el shell HBase para crear la tabla "mdds" (usando intencionalmente un nombre de familia de columna descriptivo para facilitar el seguimiento). Queremos que la familia de columnas "info" se replique en Solr, pero no los datos MOB.
El siguiente comando creará la tabla y habilitará la replicación en una familia de columnas llamada "info". Es crucial especificar la opción REPLICATION_SCOPE => '1' , de lo contrario, HBase Lily Indexer no obtendrá ninguna actualización de HBase. Queremos usar la ruta MOB en HBase para objetos de más de 10 MB. Para lograr eso, también creamos otra familia de columnas, llamada "obj", utilizando los siguientes parámetros para MOB:
IS_MOB => verdadero, MOB_THRESHOLD => 10240000
El IS_MOB El parámetro especifica si esta familia de columnas puede almacenar MOB, mientras que MOB_THRESHOLD especifica el tamaño que debe tener el objeto para que se considere un MOB. Entonces, creemos la tabla:
crear 'mdds', {NAME => 'info', DATA_BLOCK_ENCODING => 'FAST_DIFF',REPLICATION_SCOPE => '1'},{NAME => 'obj', IS_MOB => true, MOB_THRESHOLD => 10240000} Para confirmar que la tabla se creó correctamente, ejecute el siguiente comando en el shell de HBase:
hbase(main):001:0> describe 'mdds'Table mdds is ENABLEDmddsCOLUMN FAMILIES DESCRIPTION{NAME => 'info', DATA_BLOCK_ENCODING => 'FAST_DIFF', BLOOMFILTER => 'ROW', REPLICATION_SCOPE => '1' , VERSIONES => '1', COMPRESIÓN => 'NINGUNO', MIN_VERSIONS => '0', TTL => 'PARA SIEMPRE', KEEP_DELETED_CELLS => 'FALSO', BLOCKSIZE => '65536', IN_MEMORY => 'false', BLOCKCACHE => 'true'}{NAME => 'obj', DATA_BLOCK_ENCODING => 'NONE', BLOOMFILTER => 'ROW', REPLICATION_SCOPE => '0', COMPRESSION => 'NONE', VERSIONS => '1', MIN_VERSIONS => '0', TTL => 'PARA SIEMPRE', MOB_UMBRAL => '10240000', IS_MOB => 'verdadero', KEEP_DELETED_CELLS => 'FALSO', BLOCKSIZE => '65536', IN_MEMORY => 'falso', BLOCKCACHE => 'verdadero'}2 fila(s) en 0.3440 segundos Procesamiento de imágenes escaneadas con Tesseract
OCR ha recorrido un largo camino en términos de lidiar con las variaciones de fuente, el ruido de la imagen y los problemas de alineación. Aquí usaremos el motor de OCR de código abierto Tesseract, que se desarrolló originalmente como software propietario en los laboratorios de HP. Desde entonces, el desarrollo de Tesseract se ha lanzado como un software de código abierto y ha sido patrocinado por Google desde 2006.
Tesseract es una biblioteca de software altamente portátil. Utiliza la biblioteca de procesamiento de imágenes de Leptonica para generar una imagen binaria mediante la creación de umbrales adaptativos en una imagen gris o en color.
El procesamiento sigue una canalización tradicional paso a paso. El siguiente es el flujo aproximado de pasos:
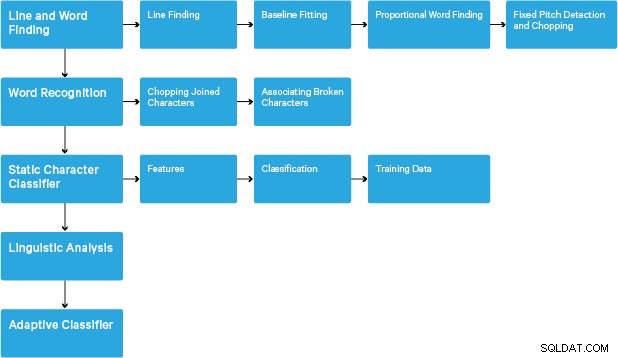
El procesamiento comienza con un análisis de componentes conectados, que da como resultado el almacenamiento de los componentes encontrados. Este paso ayuda en la inspección del anidamiento de contornos y la cantidad de contornos secundarios y nietos.
En esta etapa, los contornos se reúnen, simplemente anidando, en objetos binarios grandes (BLOB). Los BLOB se organizan en líneas de texto, y las líneas y regiones se analizan en busca de texto proporcional o de tono fijo. Las líneas de texto se dividen en palabras de manera diferente según el tipo de espaciado entre caracteres. El texto de paso fijo se corta inmediatamente por celdas de caracteres. El texto proporcional se divide en palabras usando espacios definidos y espacios difusos.
Entonces, el reconocimiento procede como un proceso de dos pasos. En la primera pasada, se intenta reconocer cada palabra por turno. Cada palabra que es satisfactoria se pasa a un clasificador adaptativo como datos de entrenamiento. El clasificador adaptativo tiene la oportunidad de reconocer con mayor precisión el texto en la parte inferior de la página. Dado que el clasificador adaptativo puede haber aprendido algo útil demasiado tarde para hacer una contribución cerca de la parte superior de la página, se ejecuta una segunda pasada por la página, en la que las palabras que no se reconocieron lo suficientemente bien se reconocen nuevamente. Una fase final resuelve los espacios borrosos y verifica hipótesis alternativas para la altura x para ubicar texto en mayúsculas pequeñas.
Tesseract en su forma actual es totalmente compatible con Unicode y está capacitado para varios idiomas. Según nuestra investigación, es una de las bibliotecas de código abierto más precisas disponibles para OCR. Como se mencionó anteriormente, Tesseract usa Leptonica. También utilizamos Ghostscript para dividir los archivos PDF en imágenes. (Puede dividirse en el formato de compresión de imágenes de su elección; elegimos PNG). Estas tres bibliotecas están escritas en C++, y para invocarlas desde programas Java/Scala, necesitamos usar implementaciones de las interfaces nativas de Java correspondientes. En nuestro trabajo, usamos los enlaces JNI de JavaPresets. (Las instrucciones de compilación se pueden encontrar a continuación). Usamos Scala para escribir el controlador Spark.
renderizador val :SimpleRenderer =nuevo SimpleRenderer( )renderer.setResolution( 300 )val images:List[Image] =renderer.render( document )
Leptonica lee las imágenes divididas del paso anterior.
ImageIO.write( x.asInstanceOf[RenderedImage], "png", imageByteStream)val pix:PIX =pixReadMem ( ByteBuffer.wrap( imageByteStream.toByteArray( ) ).array( ), ByteBuffer.wrap( imageByteStream.toByteArray( ) ).capacidad( ))
Luego usamos las llamadas a la API de Tesseract para extraer el texto. Suponemos que los documentos están en inglés aquí, por lo que el segundo parámetro del método Init es "eng".
val api:TessBaseAPI =new TessBaseAPI( )api.Init( null, "eng" )api.SetImage(pix)api.GetUTF8Text().getString()
Después de procesar las imágenes, extraemos algunos campos del texto y los enviamos a HBase.
def populateHbase ( fileName:String, lines:String, pdf:org.apache.spark.input.PortableDataStream) :Unidad ={ /** Configurar y abrir una conexión HBase */ val mddsTbl =_conn.getTable( TableName. valorDe( "mdds" )); val cf ="info" val put =new Put(Bytes.toBytes(fileName)) /** * Extraer campos aquí usando expresiones regulares * Crear objetos Put y enviarlos a HBase */ val aAndCP ="""(?s)(? m).*\d\d\d\d\d-\d\d\d\d(.*)\nRe:(\w\d\d\d\d\d\d).*"" ".r …….. las líneas coinciden { case aAndCP( addr, casenum ) => put.add( Bytes.toBytes( cf ),Bytes.toBytes( "submitter_info" ),Bytes.toBytes( addr ) ).add( Bytes .toBytes( cf ),Bytes.toBytes( "case_num" ), Bytes.toBytes( casenum )) case _ => println( "no coincidió con una expresión regular") } ……. lines.split("\n").foreach { val regNumRegex ="""Número de regulación:\s+(.+)""".r val regNameRegex ="""Nombre de regulación:\s+(.+)""" .r …….. ……. _ match { case regNumRegex(regNum) => put.add( Bytes.toBytes( cf ),Bytes.toBytes( "reg_num" ), ……. ….. case _ => print( "" ) } } put.add ( Bytes.toBytes( cf ), Bytes.toBytes( "texto"), Bytes.toBytes( líneas )) val pdfBytes =pdf.toArray.clone put.add(Bytes.toBytes( "obj"), Bytes.toBytes( " pdf" ), pdfBytes ) mddsTbl.put( poner ) …….} Si observa detenidamente el código anterior, justo antes de enviar el objeto Put a HBase, insertamos los bytes de PDF sin formato en la familia de columnas "obj" de la tabla. Usamos HBase como una capa de almacenamiento para los campos extraídos, así como la imagen sin procesar. Esto hace que sea rápido y conveniente para la aplicación extraer la imagen original, si es necesario. El código completo se puede encontrar aquí. (Vale la pena señalar que, si bien usamos las API de HBase estándar para crear objetos Put para HBase, en un sistema de producción real, sería prudente considerar el uso de las API de SparkOnHBase, que permiten actualizaciones por lotes a HBase desde Spark RDD).
Canalización de ejecución
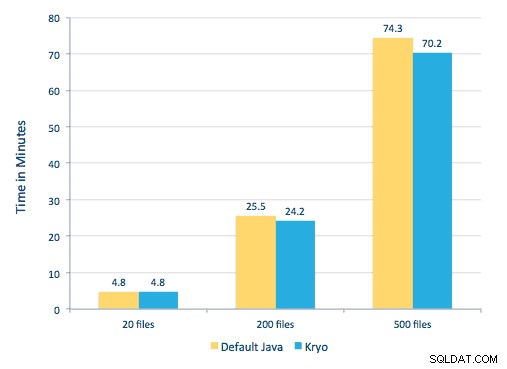
Pudimos procesar cada PDF en un marco serial. Para escalar el procesamiento, elegimos procesar estos archivos PDF de forma distribuida usando Spark. El siguiente gráfico demuestra cómo combinamos diferentes etapas de este procesamiento para convertir el flujo de trabajo en una simple llamada de macro de Spark y cargar los datos en HBase.
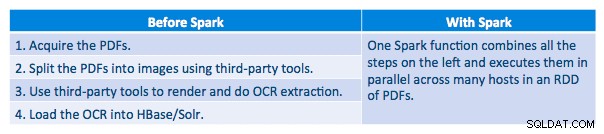
También intentamos hacer una comparación entre los métodos de serialización, pero, con nuestro conjunto de datos, no vimos una diferencia significativa en el rendimiento.
Configuración del entorno
Hardware utilizado:clúster de cinco nodos con 15 GB de memoria, 4 vCPU y 2 SSD de 40 GB
Como estábamos usando bibliotecas de C++ para el procesamiento, usamos los enlaces JNI que se pueden encontrar aquí.
Cree los enlaces JNI para Tesseract y Leptonica a partir de los ajustes preestablecidos de javaCPP:
-
- En todos los nodos:
yum -y install automake autoconf libtool zlib-devel libjpeg-devel giflib libtiff-devel libwebp libwebp-devel libicu-devel openjpeg-devel cairo-devel git clone https://github.com/bytedeco/javacpp-presets.gitcd javacpp-presets
&&sudo hacer installcd ../../../mvn clean installcd ..
- En todos los nodos:
- Construir Teseracto.
cd tesseract./cppbuild.sh install tesseractcd tesseract/cppbuild/linux-x86_64/tesseract-3.03LDFLAGS="-Wl,-rpath -Wl,/usr/local/lib" ./configuremake &&make installcd ../ ../../mvn clean installcd ..
- Cree ajustes preestablecidos de javaCPP.
mvn clean install --projects leptonica,tesseract
Usamos Ghostscript para extraer las imágenes de los PDF. Las instrucciones para compilar Ghostscript, correspondientes a las versiones de Tesseract y Leptonica utilizadas aquí, son las siguientes. (Asegúrese de que Ghostscript no esté instalado en el sistema a través del administrador de paquetes).
wget https://downloads.ghostscript.com/public/ghostscript-9.16.tar.gztar zxvf ghostscript-9.16.tar.gzcd ghostscript-9.16./autogen.sh &&./configure --prefix=/usr - -disable-compile-inits --enable-dynamicsudo make &&make soinstall &&install -v -m644 base/*.h /usr/include/ghostscript &&ln -v -s ghostscript /usr/include/ps (Dependiendo de su ldpath configuración, puede que tenga que hacerlo):sudo ln -sf /usr/lib/libgs.so /usr/local/lib/libgs.so
Asegúrese de que todas las bibliotecas necesarias estén en el classpath. Ponemos todos los frascos relevantes en un directorio llamado lib. La coma es importante a continuación:
$ para i en `ls lib/*`; exportar MY_JARS=./$i,$MY_JARS; donetesseract.jar, tesseract-linux-x86_64.jar, javacpp.jar, ghost4j-1.0.0.jar, leptonica.jar, leptonica-1.72-1.0.jar, leptonica-linux-x86_64.jar
Invocamos el programa Spark de la siguiente manera. Necesitamos especificar extraLibraryPath para las bibliotecas nativas de Ghostscript; la otra conf es necesaria para Tesseract.
spark-submit --jars $MY_JARS --num-executors 12 --executor-memory 4G --executor-cores 1 --conf spark.executor.extraLibraryPath=/usr/local/lib --confspark.executorEnv. TESSDATA_PREFIX=/home/vsingh/javacpp-presets/tesseract/cppbuild/1-x86_64/share/tessdata/ --confspark.executor.extraClassPath=/etc/hbase/conf:/opt/cloudera/parcels/CDH/lib/hbase /lib/htrace-core-3.1.0-incubating.jar --driver-class-path/etc/hbase/conf:/opt/cloudera/parcels/CDH/lib/hbase/lib/htrace-core-3.1.0 -incubating.jar --conf spark.serializer=org.apache.spark.serializer.KryoSerializer--conf spark.kryoserializer.buffer.mb=24 --class com.cloudera.sa.OCR.IdmpExtraction
Crear una colección Solr
Solr se integra perfectamente con HBase a través de Lily HBase Indexer. Para comprender cómo se realiza la integración de Lily Indexer con HBase, puede repasar nuestra publicación anterior en la sección "Comprender la replicación de HBase y Lily HBase Indexer".
A continuación, describimos los pasos que deben realizarse para crear los índices:
- Genera un archivo de configuración schema.xml de muestra:
solrctl --zk localhost:2181 instancedir --generate $HOME/solrcfg - Edite el archivo schema.xml en
$HOME/solrcfg, especificando los campos que necesitamos para nuestra colección. El archivo completo se puede encontrar aquí. - Cargue las configuraciones de Solr en ZooKeeper:
solrctl --zk localhost:2181/solr instancedir --create mdds_collection $HOME/solrcfg - Generar la colección Solr con 2 fragmentos (-s 2) y 2 réplicas (-r 2):
solrctl --zk localhost:2181/solr --solr localhost:8983/solr collection --create mdds_collection -s 2 -r 2
En el comando anterior creamos una colección Solr con dos fragmentos (-s 2) y dos réplicas (-r 2) parámetros. Los parámetros fueron suficientes para nuestro corpus, pero en una implementación real, uno tendría que establecer el número en función de otras consideraciones fuera de nuestro alcance de discusión aquí.
Registrar el indexador
Este paso es necesario para agregar y configurar el indexador y la replicación de HBase. El siguiente comando actualizará ZooKeeper y agregará mdds_indexer como par de replicación para HBase. También insertará configuraciones en ZooKeeper, que Lily HBase Indexer usará para apuntar a la colección correcta en Solr. |
hbase-indexer add-indexer -n mdds_indexer -c indexer-config.xml -cp solr.zk=localhost:2181/solr -cp solr.collection=mdds_collection.
Argumentos:
-n mdds_indexer– especifica el nombre del indexador que se registrará en ZooKeeper-c indexer-config.xml– archivo de configuración que especificará el comportamiento del indexador-cp solr.zk=localhost:2181/solr– especifica la ubicación de la configuración de ZooKeeper y Solr. Esto debe actualizarse con la ubicación específica del entorno de ZooKeeper.-cp solr.collection=mdds_collection– especifica qué colección actualizar. Recuerde el paso de Configuración de Solr donde creamos la colección 1.
El index-config.xml el archivo es relativamente sencillo en este caso; todo lo que hace es especificar al indexador qué tabla mirar, la clase que se usará como mapeador (com.ngdata.hbaseindexer.morphline.MorphlineResultToSolrMapper ) y la ubicación del archivo de configuración de Morphline. De forma predeterminada, el tipo de asignación se establece en fila , en cuyo caso el documento Solr se convierte en la fila completa. Param name="morphlineFile" especifica la ubicación del archivo de configuración Morphlines. La ubicación podría ser una ruta absoluta de su archivo Morphlines, pero dado que está utilizando Cloudera Manager, especifique la ruta relativa como morphlines.conf.
El contenido del archivo de configuración de hbase-indexer se puede encontrar aquí.
Configurar e iniciar el indexador Lily HBase
Cuando habilita Lily HBase Indexer, debe especificar la lógica de transformación de Morphlines que permitirá que este indexador analice las actualizaciones de la tabla de dispositivos médicos y extraiga todos los campos relevantes. Vaya a Servicios y elija Lily HBase Indexer que agregó anteriormente. Seleccione Configuraciones->Ver y editar->Todo el servicio->Morphlines . Copie y pegue el archivo Morphlines.
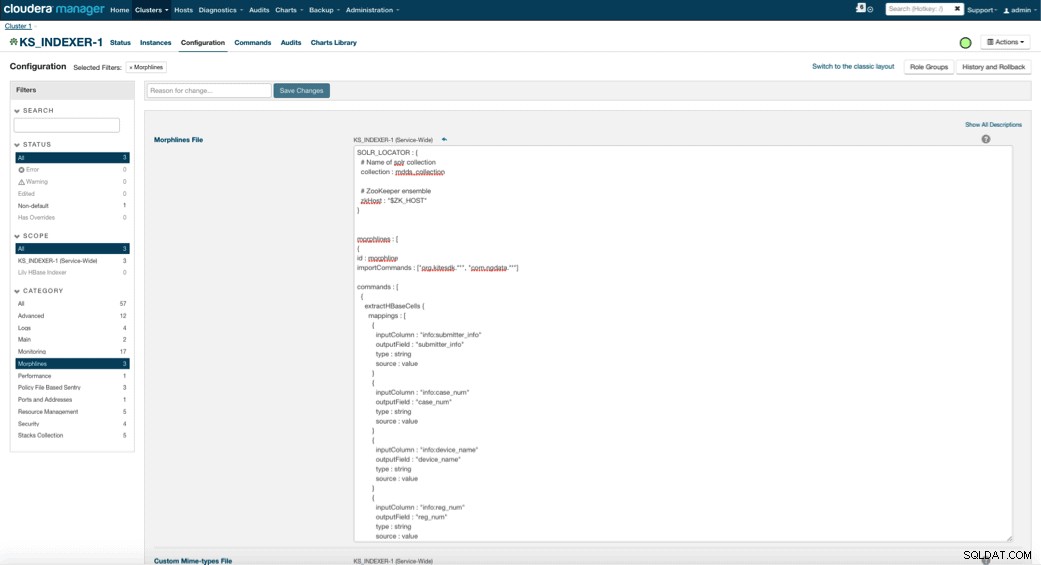
La biblioteca de morphlines de dispositivos médicos realizará las siguientes acciones:
- Lea los eventos de correo electrónico de HBase con
extractHBaseCellscomando - Convierta la fecha/marca de tiempo en un campo que Solr entienda, con
convertTimestampcomandos - Elimine todos los campos adicionales que no especificamos en schema.xml, con
sanitizeUknownSolrFieldscomando
Descargue una copia de este archivo Morphlines desde aquí.
Una nota importante es que Lily HBase Indexer generará automáticamente el campo de identificación. Esa configuración se puede configurar en el archivo index-config.xml anterior especificando el atributo de campo de clave única. Es una buena práctica dejar el nombre predeterminado de id, ya que no se especificó en el archivo xml anterior, se generó el campo de id predeterminado y será una combinación de RowID.
Acceso a los datos
Tiene la opción de muchas herramientas visuales para acceder a las imágenes indexadas. HUE y Solr GUI son muy buenas opciones. HBase también permite una serie de técnicas de acceso, no solo desde una GUI, sino también a través del shell de HBase, la API e incluso técnicas de secuencias de comandos simples.
La integración con Solr le brinda una gran flexibilidad y también puede proporcionar opciones de búsqueda muy simples y avanzadas para sus datos. Por ejemplo, configurar el archivo schema.xml de Solr de modo que todos los campos dentro del objeto de correo electrónico se almacenen en Solr permite a los usuarios acceder al cuerpo completo del mensaje a través de una búsqueda simple, con el compromiso de espacio de almacenamiento y complejidad informática. Alternativamente, puede configurar Solr para almacenar solo una cantidad limitada de campos, como la identificación. Con estos elementos, los usuarios pueden buscar rápidamente en Solr y recuperar el ID de fila que, a su vez, se puede usar para recuperar campos individuales o la imagen completa de HBase.
El ejemplo anterior almacena solo el ID de fila en Solr pero indexa todos los campos extraídos de la imagen. La búsqueda de Solr en este escenario recupera los ID de fila de HBase, que luego puede usar para consultar HBase. Este tipo de configuración es ideal para Solr, ya que mantiene bajos los costos de almacenamiento y aprovecha al máximo las capacidades de indexación de Solr.
Consultas de muestra
A continuación se muestran algunos ejemplos de consultas que se pueden realizar desde la aplicación en Solr. La idea es que el cliente consultará inicialmente los índices de Solr, devolviendo el ID de fila de HBase. Luego consulte HBase para el resto de los campos y/o la imagen sin procesar original.
- Dame todos los documentos que se archivaron entre las siguientes fechas:
https://hbase-solr2-1.vpc.cloudera.com:8983/solr/mdds_collection/select?q=received:[2010-01 -06T23:59:59.999Z HASTA 2010-02-06T23:59:59.999Z]
- Dame los documentos que se archivaron bajo el nombre normativo de los sistemas de rayos X móviles:
https://hbase-solr2-1.vpc.cloudera.com:8983/solr/mdds_collection/select?q=reg_name:Mobile sistema de rayos x
- Dame todos los documentos que fueron archivados por fabricantes chinos:
https://hbase-solr2-1.vpc.cloudera.com:8983/solr/mdds_collection/select?q=submitter_info:*China*
Los ID de los documentos de Solr son los ID de fila en HBase; la segunda parte de la consulta será a HBase para extraer los datos (incluido el PDF sin procesar, si es necesario).
Acceso a través de HUE
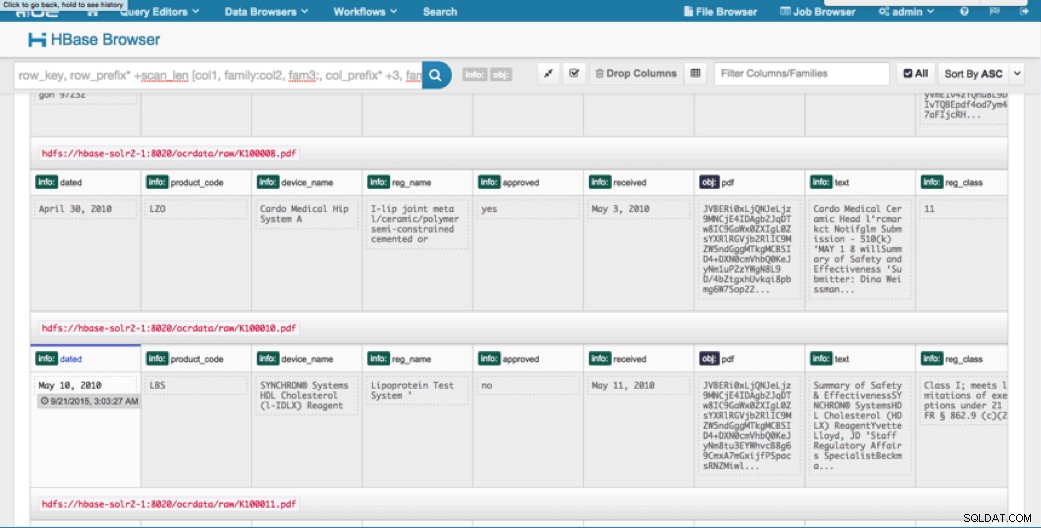
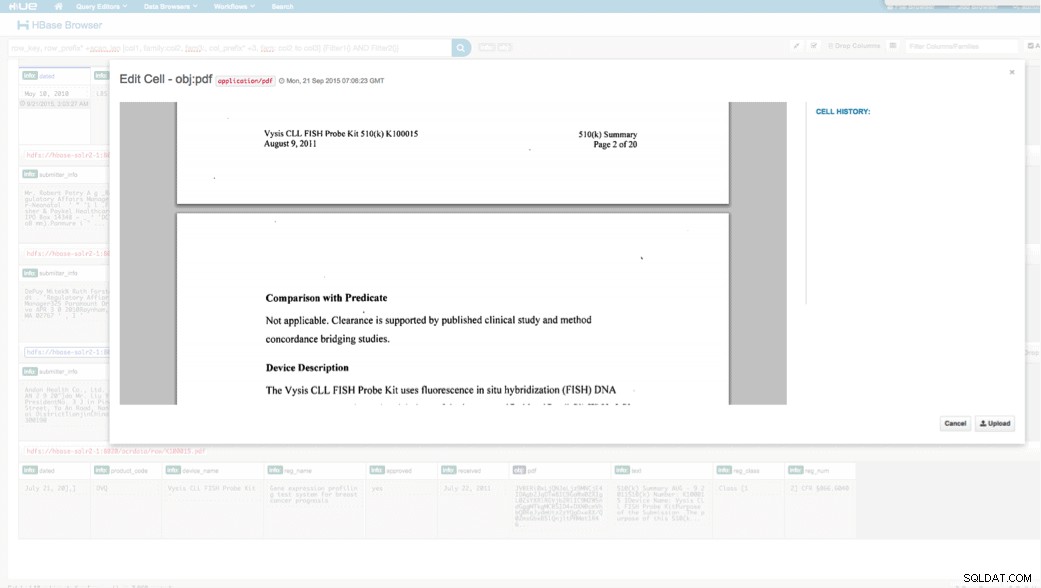
Podemos ver los datos cargados a través del navegador HBase en HUE. Una gran ventaja de HUE es que puede detectar los archivos binarios de PDF y mostrarlos cuando se hace clic en ellos.
A continuación se muestra una instantánea de la vista de los campos analizados en las filas de HBase y también una vista renderizada de uno de los objetos PDF almacenados como MOB en la familia de columnas obj.
Conclusión
En esta publicación, hemos demostrado cómo usar tecnologías estándar de código abierto para realizar OCR en documentos escaneados usando un programa Spark escalable, almacenándolos en HBase para una recuperación rápida e indexando la información extraída en Solr. Debería ser evidente que:
- Dado el formato de especificación del mensaje, podemos extraer campos y pares de valores y hacer que se puedan buscar a través de Solr.
- Estos campos de datos pueden cumplir con los requisitos de IDMP de hacer que los datos heredados sean electrónicos, lo que entrará en vigencia el próximo año.
- Los campos, así como las imágenes sin procesar, se pueden conservar en HBase y se puede acceder a ellos a través de las API estándar.
Si necesita procesar documentos escaneados y combinar los datos con otras fuentes en su empresa, considere usar una combinación de Spark, HBase, Solr, junto con Tesseract y Leptonica. ¡Puede ahorrarle una cantidad considerable de tiempo y dinero!
Jeff Shmain es arquitecto sénior de soluciones en Cloudera. Tiene más de 16 años de experiencia en la industria financiera con una sólida comprensión del comercio de valores, el riesgo y las regulaciones. En los últimos años, ha trabajado en varias implementaciones de casos de uso en 8 de los 10 bancos de inversión más grandes del mundo.
Vartika Singh es consultor sénior de soluciones en Cloudera. Tiene más de 12 años de experiencia en aprendizaje automático aplicado y desarrollo de software.